日前,深圳华大生命科学研究院邹远强/肖亮团队,依托国家超级计算郑州中心的强大计算和存储资源,完成肠道培养菌基因组与全球10000+健康人的肠道宏基因组队列序列比对和分析,提出新的可培养人类肠道细菌参考基因组的基因组图谱,助力肠道微生物的研究与转化应用。相关研究成果以《The genomic landscape of reference genomes of cultivated human gut bacteria》为题发表在国际学术期刊《自然·通讯》(Nature Communications),并对国家超级计算郑州中心致谢。
该研究构建了3324个菌株的高质量基因组草图,为研究者们全面认识人肠道细菌参与膳食纤维代谢及短链脂肪酸合成的过程、发现具有潜在价值的次级代谢产物合成基因簇、构建肠道噬菌体-细菌相互作用网络提供帮助。这些细菌基因组和菌株将是人体共生微生物研究的宝贵资源。
人体是一个由人体细胞和所有共生微生物细胞构成的“超级生物体”,人体肠道定殖着复杂多样的共生微生物,包括细菌、古细菌、真菌和病毒。随着肠道微生物研究的深入,越来越多新的微生物序列通过宏基因组测序被发现,但能够成功获取的纯培养菌株只是其中一小部分。此外,高质量参考基因组数据的缺乏也阻碍了更高物种分辨率下的宏基因组研究。
本项目通过新采集144例中国健康志愿者的样本,结合已公开发表的CGR资源库进行分析。使用近40种培养条件共获得2万多株纯培养菌株,选择其中4066株代表性菌株进行基因组测序和组装,最终获得3324个高质量基因组。其中包含的基因组数据涵盖人体肠道微生物中8个门的527个物种,包括179个尚未被报道的细菌新物种。与目前已发表的肠道微生物基因组数据集相比,CGR2提供了189个特有的物种,进一步填补了培养组领域数据与资源的空白。
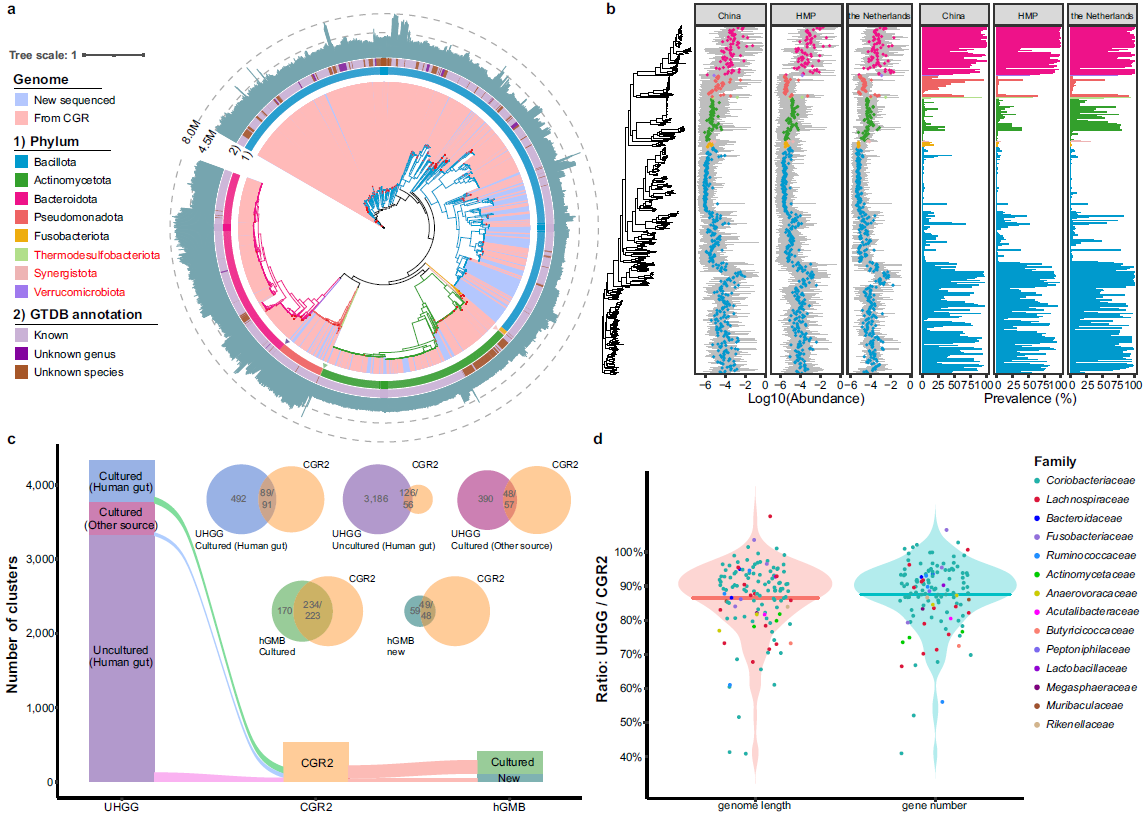
图1:肠道培养菌的分类和在人群中的分布(图源自Nature Communications)
研究团队培养的双歧杆菌也有完整的母乳寡糖代谢途径,进一步支持了母乳寡糖可作为益生元在人体肠道中通过为双歧杆菌提供养料调节肠道菌群,进而帮助改善人体健康。研究描绘了肠道细菌参与碳水化合物代谢的功能图谱,发现近200个菌株具有完整的膳食纤维(包括果胶、纤维素和菊粉)代谢及短链脂肪酸的合成通路,这些膳食纤维对人体的健康有着重要的作用。研究发现24种类型的4000多个次级代谢物合成基因簇,表明肠道微生物同样具有丰富多样的次级代谢产物合成潜力,可作为抗菌肽、蛋白酶抑制剂等活性物质的来源。研究预测到2820条高质量噬菌体序列,其中绝大部分未被报道,但CGR2能提供这些噬菌体明确的宿主信息,并揭示其与宿主间复杂的作用关系,为深入探究噬菌体与人体健康之间的关系和基于噬菌体对疾病的靶向治疗提供了重要参考。
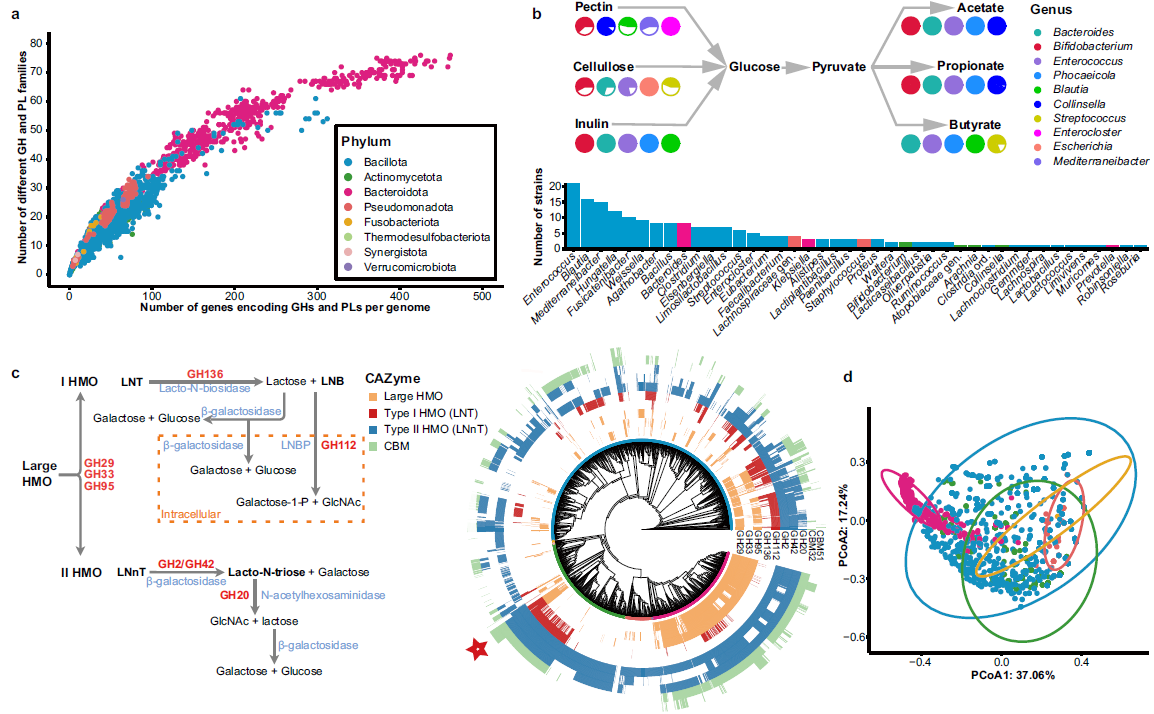
图2:碳水化合物代谢基因分布(图源自Nature Communications)
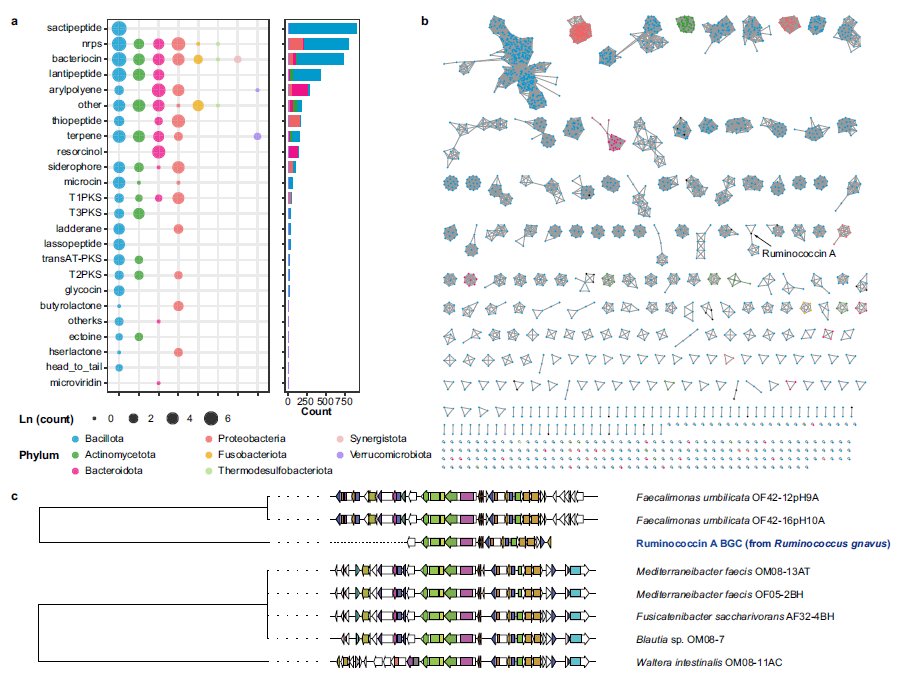
图3:次级代谢产物合成基因丰度(图源自Nature Communications)
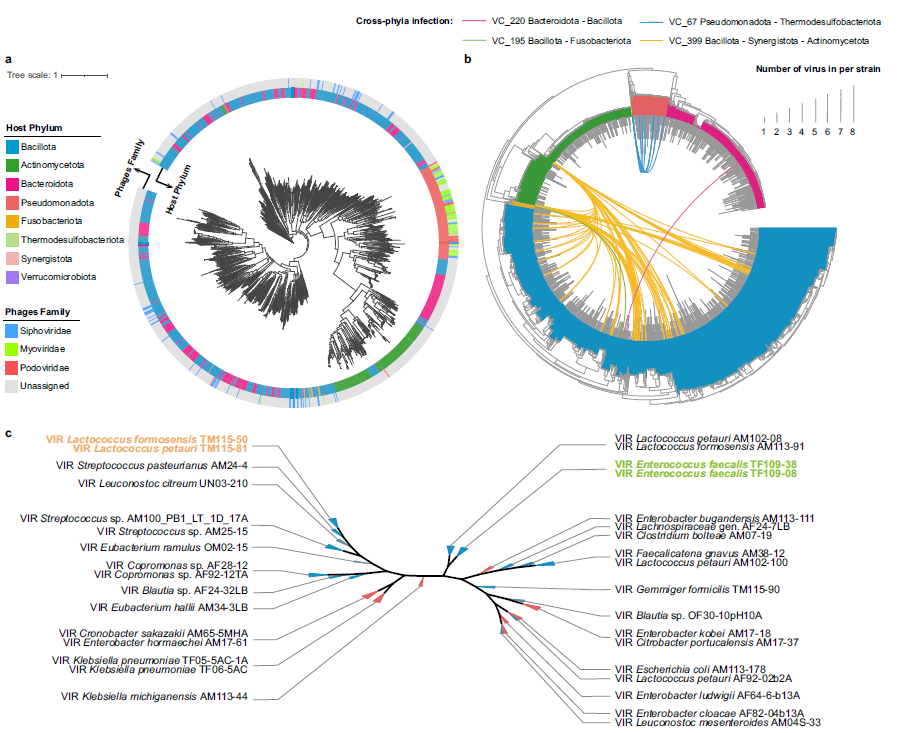
图4:噬菌体分类和与宿主互作(图源自Nature Communications)
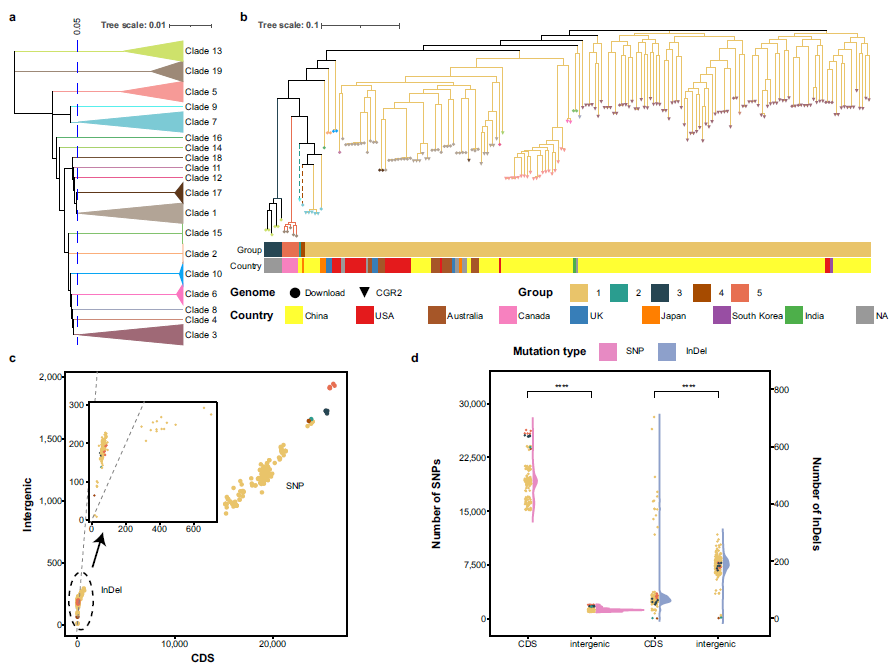
图5:Collinsella aerofaciens全基因组关联分析(图源自Nature Communications)